Cette fois, c’est la bonne! Je vais t’expliquer ce que ça veut dire, ces représentations de protéines avec des rubans⬇️⬇️⬇️. Comme ça, tu sauras pourquoi on n’y voit pas d’atomes. Tu vas voir, c’est une question de simplification…
Les protéines ce sont des macromolécules présentes chez tous les êtres vivants. Des molécules géantes. Et comme toutes les macromolécules, elles sont construites avec des briques de base plus petites. Là, ce sont les acides aminés.
Pour construire des protéines, il faut enchaîner des acides aminés (AA), selon une suite (une séquence) précise, et ça, c’est un processus cellulaire complexe —la traduction— qui le permet. Mais on ne va pas parler de traduction, c’est trop pour ce thread.
Faut juste que tu saches que selon la séquence en AA d’une protéine, ça détermine la manière dont l’assemblage se conforme en 3D (càd se structure en trois dimensions, pour être plus clair). C’est donc important de voir comment on enchaîne les AA.
Pour comprendre comment les AA peuvent être enchaînés, tu ne vas pas pourvoir faire l’économie de quelque bases de chimie⬇️⬇️⬇️. Et d’ailleurs, pour comprendre ce qu’est un acide aminé, c’est pareil. Si tu t’y connais déjà, tu peux passer à la suite directement.
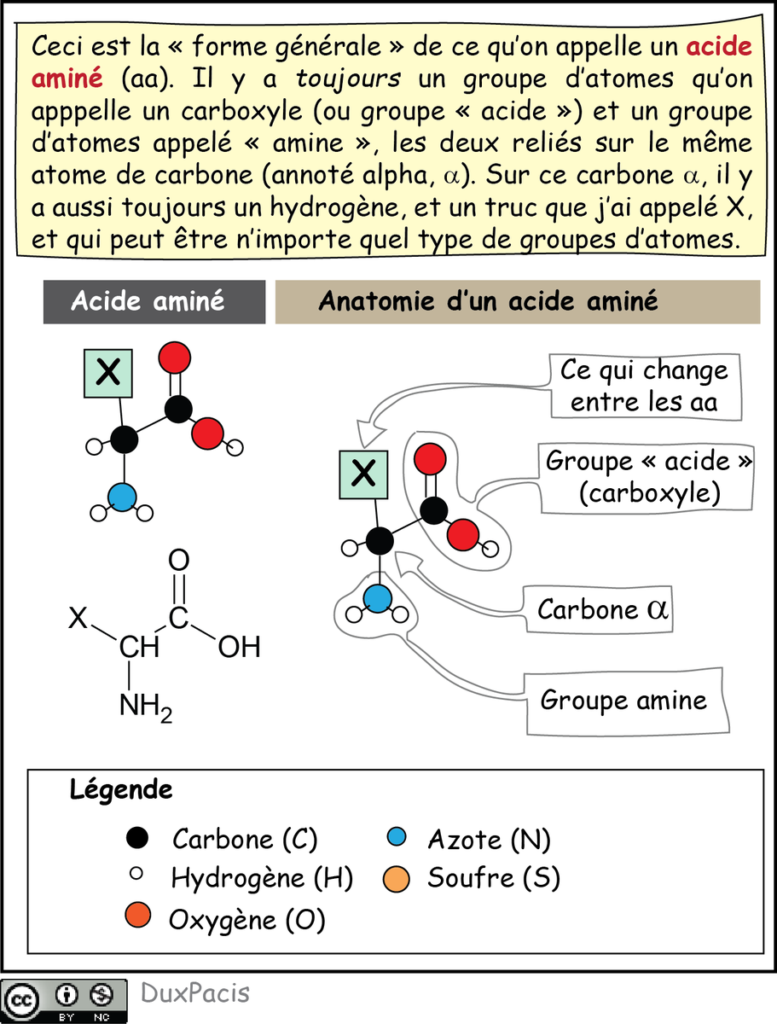
Les AA sont de petites molécules qui contiennent TOUJOURS deux groupes chimiques⬇️⬇️: un qui s’appelle carboxyle (COOH) (qui est un groupe *acide* pour des raisons que je ne vais pas te détailler ici) et l’autre qui s’appelle amine (NH2), d’où le nom acide aminé, tu suis?

Ils ont tous un point commun: le carboxyle et l’amine sont sur le MÊME carbone central. Tu vas voir que ce sont ceux deux trucs qui vont jouer le rôle de « colle » pour enchaîner les acides aminés entre eux comme des Legos.
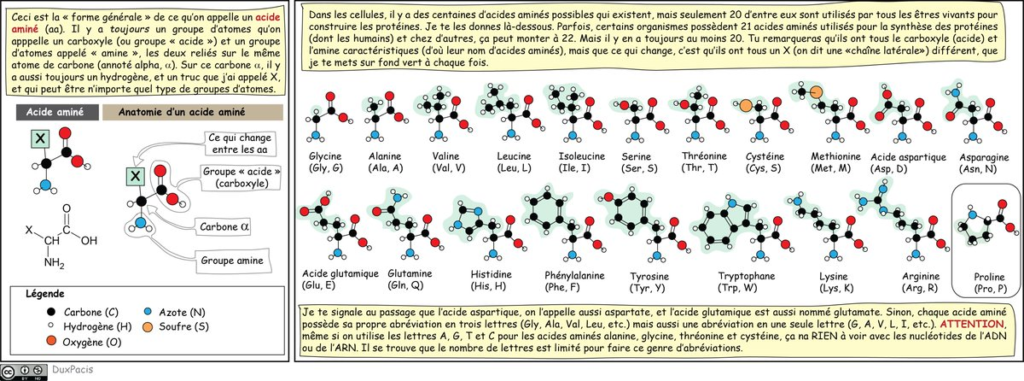
Ce qui change entre les 20 acides aminés utilisés pour construire les protéines, c’est ce qu’on appelle leur « chaîne latérale ». Il y 20 chaînes latérales différentes, donc 20 acides aminés. Logique.
Je te donne la liste des 20 acides aminés là-dessous⬇️⬇️⬇️, avec leurs noms, et les abréviations qu’on utilise pour les symboliser ; en trois lettres (par ex. Glu, Asp, Trp, etc.) ou en une seule lettre (E, D, W, etc.). Les détails sont là-dedans si ça t’intéresse
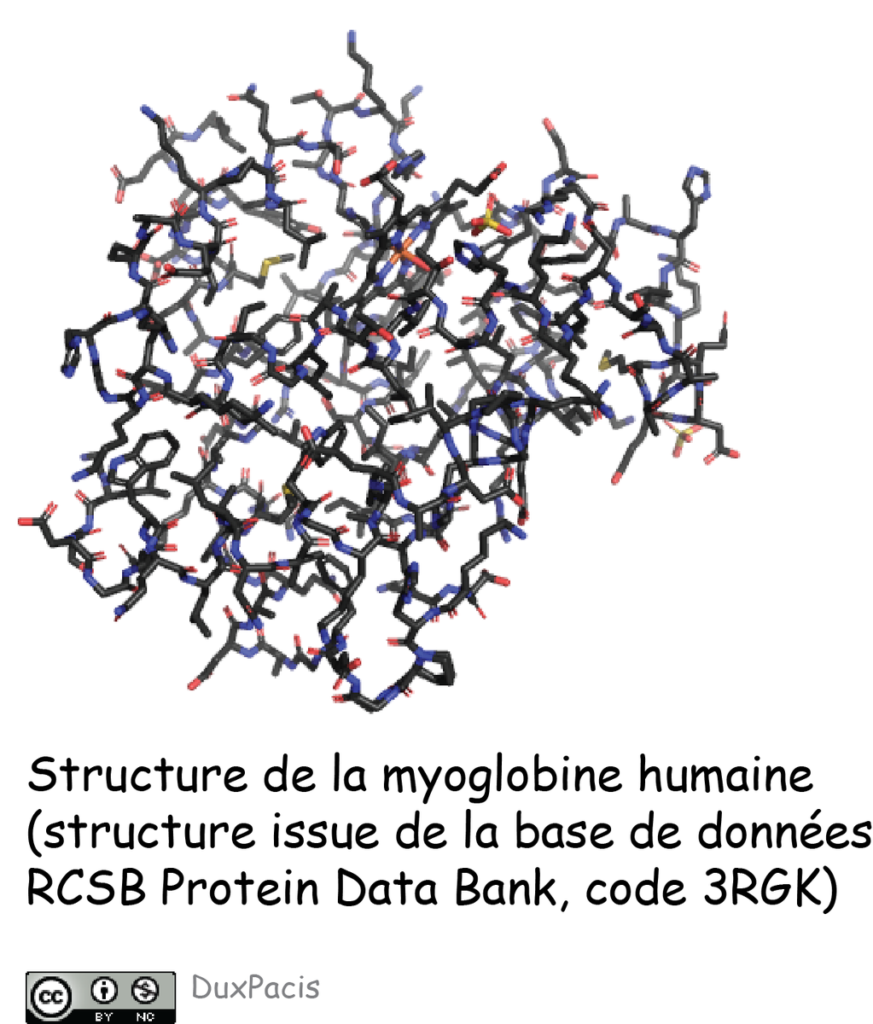
Pour faire une protéine particulière, il faut relier des acides aminés entre eux selon un ordre précis (sa séquence). Je te donne l’exemple d’une « petite » protéine appelée myoglobine⬇️⬇️⬇️, constituée de 154 acides aminés.

Pour enchaîner, il faut relier un AA par son carboxyle avec un second AA par son amine. Le résultat, c’est ce qu’on appelle une « liaison peptidique »⬇️⬇️⬇️. Le carboxyle et l’amine n’existent alors plus en tant que tels, mais deviennent la liaison peptidique.
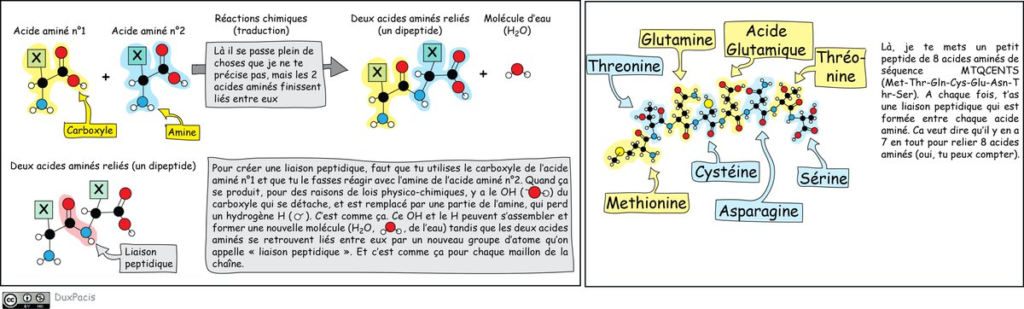
Du coup, tu comprends que quand tu enchaînes plusieurs acides aminés, tu as plusieurs « liaisons peptidiques » et que c’est donc un polyPEPTIDE. Quand ça devient grand, on parle de protéine.
Ce sont toujours des enchaînements linéaires d’AA (les uns à la suite des autres). Mais ATTENTION, la protéine n’est pas pour autant une corde linéaire. Quand elle est assemblée, elle s’organise dans l’espace (en 3D) au gré des interactions entre les acides aminés de la chaîne. Parce que oui, y a un truc que je t’ai pas dit. Les AA ont tous —à cause de leurs chaînes latérales différentes— des propriétés physicochimiques différentes.
Y en a qui, au pH de la cellule, sont chargés négativement (Glu, Asp), d’autres positivement (Lys, Arg). C’est dû aux propriétés chimiques des carboxyles et des amines des chaînes latérales⬇️ (pas ceux qui servent à faire les liaisons peptidiques, qui n’existent plus !)
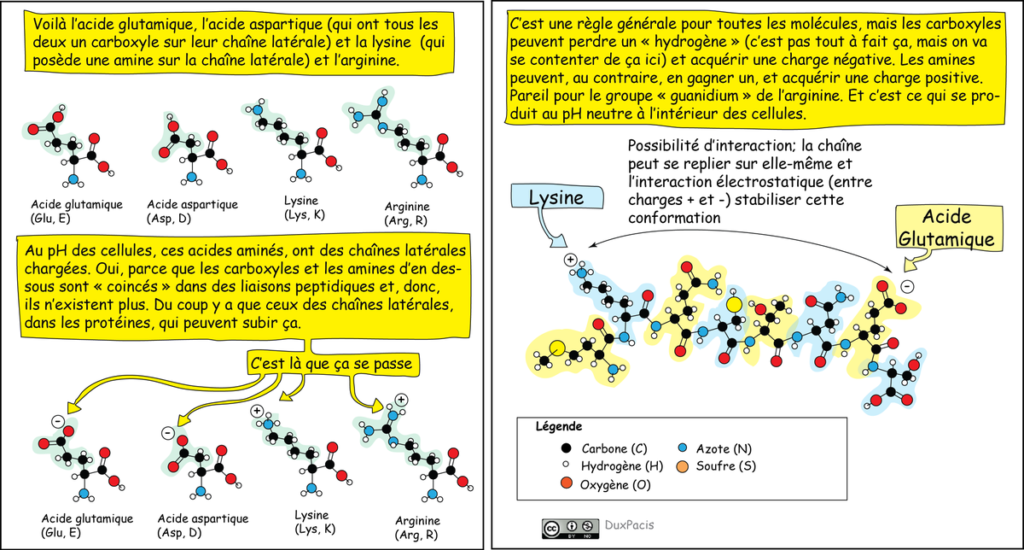
Y en a qui sont hydrophobes (« n’aiment pas l’eau », càd « ne peuvent pas interagir avec elle »), d’autres qui sont hydrophiles (« aiment l’eau » càd « peuvent interagir avec elle). Ces différences seraient longues à expliquer.
Les AA hydrophobes ont tendance à se rassembler, formant des « cœurs hydrophobes », les AA chargés positivement à interagir avec ceux chargés négativement, etc. La chaîne d’AA, donc, se replie au gré de ces interactions, et adopte une forme en 3D qu’on appelle son REPLIEMENT. La séquence de la myoglobine, càd l’ordre d’enchaînement des AA et les interactions qu’ils forment entre eux, influence son repliement, et elle adopte ce repliement-là⬇️⬇️⬇️
[BONUS] Du coup, je t’ai fait une animation pour que tu voies la myoglobine en 3D avec la même représentation. Je l’ai faite avec le fichier de la structure de la PDB (lien : rcsb.org/structure/3RGK) et le logiciel PyMol (lien : pymol.org/2/).
Et là, tu vas me dire : « on ne voit rien ! ». Oui, et c’est bien le souci! Du coup, pour simplifier la visualisation de la structure 3D (le repliement) d’une protéine, les biologistes ont inventé plusieurs représentations différentes⬇️⬇️⬇️.
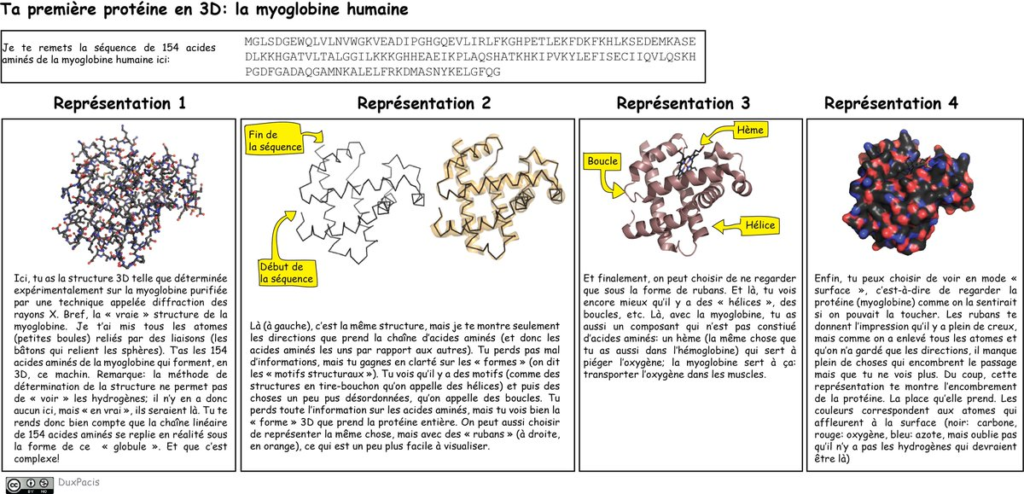
En fait, pour qu’une chaîne d’AA se replie en 3D, ça implique que les liens entre les AA soient flexibles et qu’ils s’orientent les uns par rapport aux autres de différentes manières. Les représentations « en rubans » te disent, justement, comment ça se passe.
Si les AA d’une chaîne forment un assemblage linéaire, bien droit, on dit que c’est un « brin bêta », et on le représente par un ruban avec une flèche (qui indique le sens de la séquence)⬇️⬇️⬇️. Tu vois plus les AA ni les atomes, juste leur direction générale.

Si, en vertu de leurs interactions, les AA forment un « serpentin » en tire-bouchon (on appelle ça une hélice alpha), on représente ça par un ruban en hélice⬇️⬇️⬇️. Là non plus, tu vois plus les AA, ni les atomes, mais tu sais que la structure est hélicoïdale.

S’il n’y a pas de direction privilégiée ou de structure particulière, on représente ça par des filaments qu’on appelle des « boucles ». Ca te dit qu’il n’y a pas de brin bêta ou d’hélice alpha, mais des trucs souvent flexibles, qui peuvent bouger.
Quel intérêt? C’est plus facile de comparer la structure de deux protéines différentes, et savoir si elles ont des similarités de structure, même si elles n’ont pas la même séquence. Regarde la myoglobine humaine et celle du cachalot (ouais, pourquoi pas ??)⬇️⬇️

Tu vois qu’elles n’ont pas exactement la même séquence. Pourtant, leurs deux structures sont quasiment identiques (du premier coup d’œil qd t’as l’habitude)⬇️. Les changements de séquence n’ont pas changé la structure = compatibles avec la subsistance du même repliement.
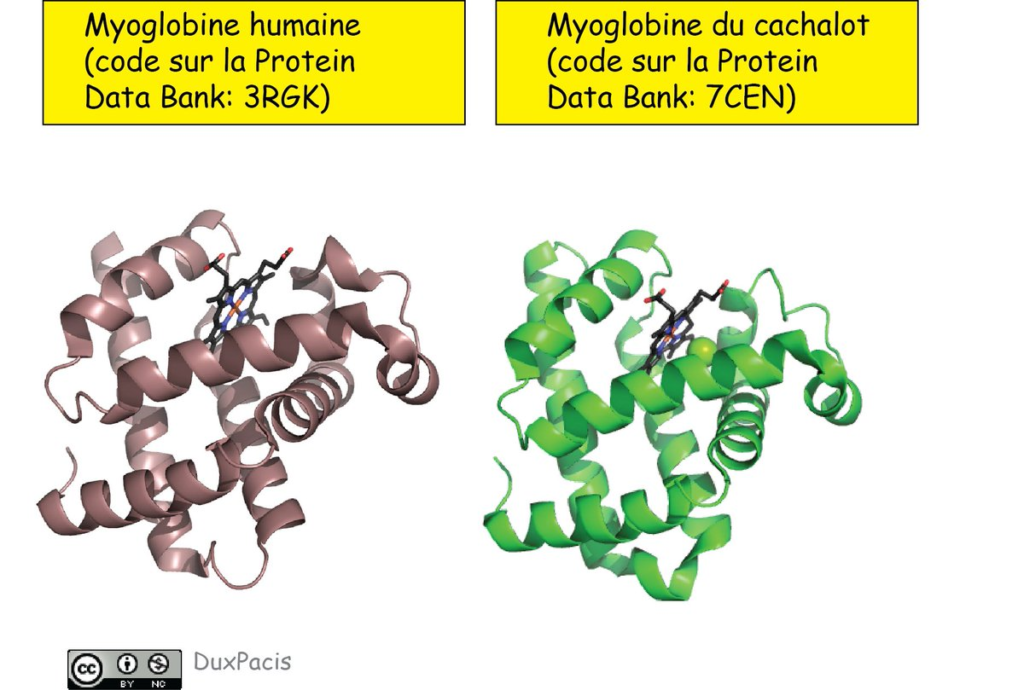
Et comme la structure d’une protéine influence fortement la fonction qu’elle peut avoir dans la cellule, les deux sont toujours des myoglobines, avec la même fonction de transport de l’oxygène, même avec des séquences qui ne sont pas identiques.
Sans la représentation en rubans, ç’aurait été beaucoup plus difficile à voir. Tu comprends mieux pourquoi on l’utilise un peu partout. Tu peux d’ailleurs voir que l’hémoglobine (une autre protéine) a la même structure sans avoir (exactement) la même séquence⬇️
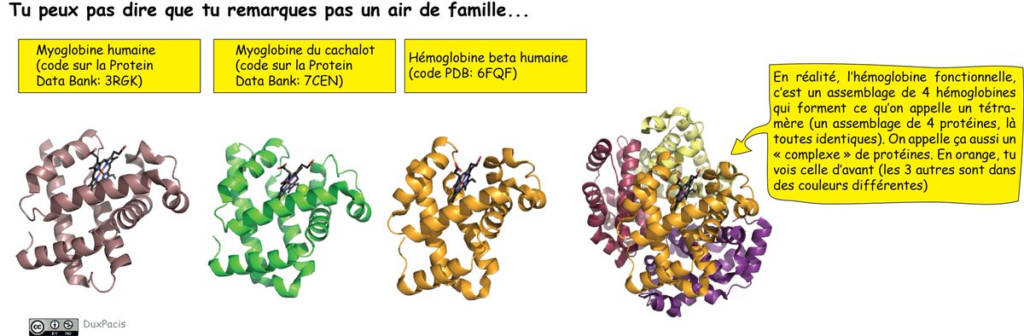
Mais sa fonction est similaire: transporter l’oxygène (dans le sang, alors que la myoglobine, c’était dans les muscles). Bref, tu vois que les fonctions similaires sont liées à des structures similaires. Et avec ce genre de représentations, ça se voit aussi.
J’espère que ça t’aura éclairé. C’était dense ! La prochaine fois, on verra d’autres choses. On parlera même de la protéine Spike, parce qu’il n’y a pas de raison qu’on ne choisisse pas un exemple qui parle à tout le monde.
[CONCLUSION]
Maintenant t’es passé.e du côté obscur: tu sais mieux pourquoi on représente les protéines par des rubans, ça sera très utile dans d’autres contextes. Mais fallait bien parler de ça pour lever le voile de flou artistique qui reposait sur les protéines. On se bientôt!
Originally tweeted by DuXpa6 (@duxpacis) on 28 November 2021.
